浅谈DNA甲基化检测技术的优缺点
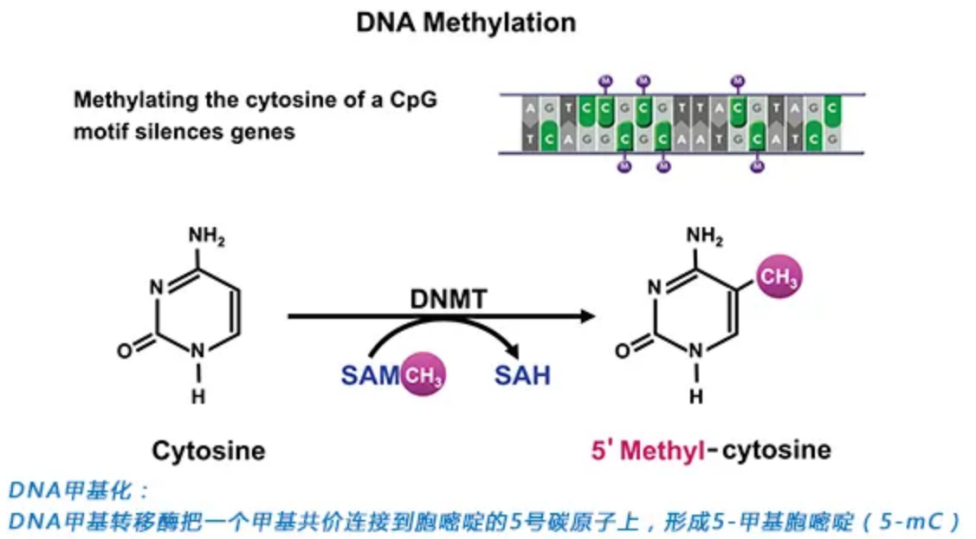
DNA甲基化(DNA methylation)为DNA化学修饰的一种形式,能够在不改变DNA序列的前提下,改变遗传表现。所谓DNA甲基化是指在DNA甲基化转移酶的作用下,在基因组CpG二核苷酸的胞嘧啶5'碳位共价键结合一个甲基基团。大量研究表明,DNA甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达。DNA甲基化在干细胞维持和自我更新与分化,个体的衰老和发育异常及疾病(如肿瘤、糖尿病、精神病及神经系统疾病等复杂疾病)的发生发展中起着决定性的作用。
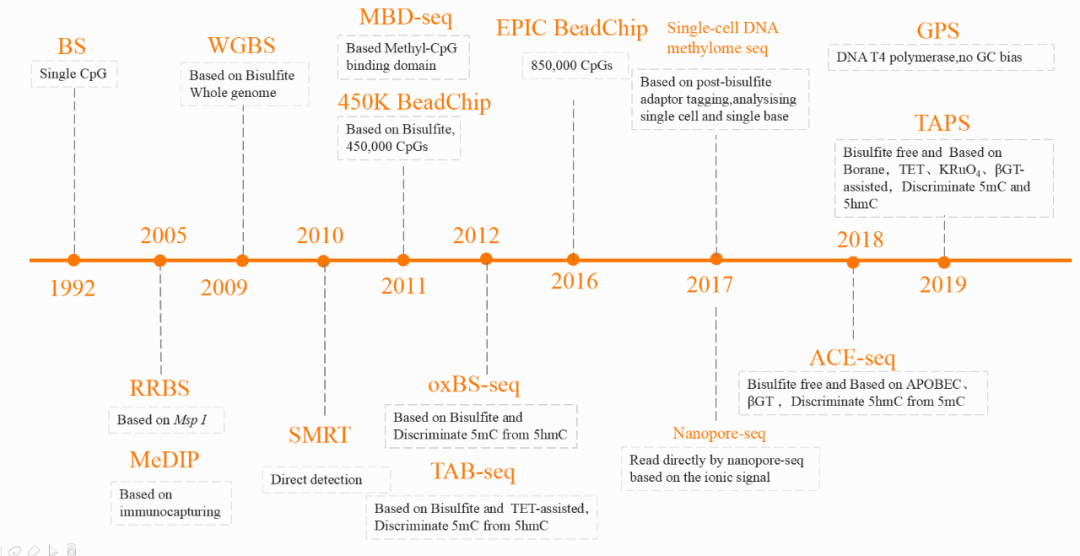
随着高通量测序技术的发展,我们能够从全基因组水平来分析5’-甲基胞嘧啶,这就是 “DNA甲基化测序”!且近年来测序成本的不断下降及测序技术的迭代更新,DNA甲基化测序方法可选择性更多了。
目前表观遗传学DNA甲基化研究测序方法常见的有:全基因组重亚硫酸盐甲基化测序[WGBS]、简化甲基化测序[RRBS]、甲基化DNA免疫共沉淀测序[MeDIP-seq]、甲基化捕获测序[Methyl-Seq]、甲基化芯片检测[EPIC2.0]等5种。
全基因组重亚硫酸盐甲基化测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平。可以检测全基因组所有位点的甲基化水平;是一种时间比较早,比较通用的检测技术。
常规全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T,进而区分该碱基胞嘧啶C是否发生了甲基化。
技术优势:
💠 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息;
💠 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。
技术缺陷:
💠 价格昂贵,性价比不高,不适合做大量样本;
💠 测序深度低,人的样本,90G数据量平均深度在12-15X。
简化甲基化测序(RRBS)
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集CpG位点相对比较集中的区域并进行重亚硫酸盐测序。该技术提高了高CpG区域的测序深度,主要在CpG岛、区域可以获得高精度的分辨率。
技术优势:
💠 单碱基分辨率:在其覆盖范围内可达到单碱基分辨率;
💠 性价比高:测序区域针对高CpG区域,数据利用率更高。
技术缺陷:
💠 重复性低:技术的重复性大概只有85%-95%;
💠 测序深度:10g的数据量,平均测序深度30X,测序深度大于30X的位点,占比比较低。
甲基化DNA免疫共沉淀测序MeDIP-seq
MeDIP-Seq(Methylated DNA Immunoprecipitation Sequencing)测序是基于抗体富集原理进行测序的全基因组甲基化检测技术,采用甲基化DNA免疫共沉淀技术,通过5'-甲基胞嘧啶抗体特异性富集 基因组上发生甲基化的DNA片段,然后通过高通量测序可以在全基因组水平上进行高精度的CpG密集的高甲基化区域研究。
技术优势:
💠 成本低:大大降低了测序数据量(每个样本数据只需1-2G),测序成本低;
技术缺陷:
💠 单碱基分辨:无法检测单个位点的甲基化值,分辨率在50-100bp。
靶向甲基化测序Methyl-Seq
Methyl-Seq(Methylated DNA Target Sequencing)测序是探针捕获原理进行测序的全基因组甲基化检测技术,实验原理有点类似全外显子测序,然后通过高通量测序可以在全基因组水平上进行高精度的CpG密集的高甲基化区域研究,设计区间在全基因组水平CpG islands、CpG island shores、CpG island shelves、 undermethylated 区域、启动子、肿瘤及组织特异性甲基化区域 (DMR)、增强子、Ensemble 调控区域和DNase I 高敏感位点。
技术优势:
💠 单碱基分辨率:在其覆盖范围内可达到单碱基分辨率;
💠 测序深度:数据量测到15G可达到80-100x的测序深度,性价比高;
💠 重复性高:技术重复相关性R2>0.97;
💠 可以进行甲基化单倍型分析;
💠 甲基化状态不受snp位点的影响;
💠 可根据客户需求灵活设计不同的panel。
技术缺陷:
💠 目前产品化的靶向甲基化测序产品只能检测人、大鼠、小鼠。
illumina 甲基化芯片检测
illumina 甲基化芯片(EPIC)是基于芯片杂交捕获单碱基延伸的原理设计的一款芯片,目前已经升级从最开始的27K、450K、850K升级到935K芯片,芯片也通过不断的改进,完善升级到目前认为最优的版本,甲基化芯片也是全基因组覆盖的一款产品,全面覆盖CpG岛、启动子、编码区、 开放染色质和增强子。此外还包括CpG岛外的CpG位点,已知DMR位点,脱氧核糖核酸酶超敏位点以及miRNA启动子区域。
技术优势:
💠 单碱基分辨率:在其覆盖范围内可达到单碱基分辨率;
💠 重复性高:技术重复相关性R2>0.99;
💠 有大量的公共数据库可以利用。
技术缺陷:
💠 不适合甲基化区段的分析。
最后小编说一下自己对这几个技术的看法,仅代表个人看法,无利益倾向。
在这几种技术中MeDIP-Seq和RRBS都是至少十几年的技术了,小编认为目前出现的新的技术已经把这些技术慢慢替代了。
WGBS虽然也很久了,这个技术在位点覆盖程度上是无可替代的,比如一些内含子区域的甲基化。只不过是拿到这些位点的甲基化信息是否对研究真的有意义。以及低深度测序带来的数据准确性的问题,是个需要思考的问题。
935K芯片和甲基化捕获测序,个人认为是我们目前研究甲基化最好技术手段。935K芯片经过450K和850K芯片这些版本优化,在稳定性上是现有技术无法媲美的。甲基化捕获测序我个人觉得就像WGS发展成WES一样。在提高了测序深度的同时,节约测序成本,这点在大队列研究中极为重要。另外就是甲基化捕获测序后期可以进行定制化捕获测序,可以根据前期筛选的位点进行定制,这一点是现有技术都做不到。尤其在临床转化上优势极为明显。
本文转载自微信订阅号“中科普瑞科研服务”,如有侵权,请联系弘德网删除。